Glucose-6-phosphate déshydrogénase
Apparence
| Glucose-6-phosphate déshydrogénase | ||
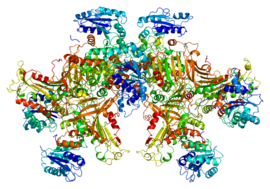 Structure d'une G6PD humaine cristallisée (PDB 1QKI[1]) | ||
| Caractéristiques générales | ||
|---|---|---|
| Synonymes | G6PD | |
| N° EC | 1.1.1.49 | |
| Homo sapiens | ||
| Locus | Xq28 | |
| Masse moléculaire | 59 257 Da[2] | |
| Nombre de résidus | 515 acides aminés[2] | |
| Entrez | 2539 | |
| HUGO | 4057 | |
| OMIM | 305900 | |
| UniProt | P11413 | |
| RefSeq (ARNm) | NM_000402.4, NM_001042351.2 | |
| RefSeq (protéine) | NP_000393.4, NP_001035810.1 | |
| Ensembl | ENSG00000160211 | |
| PDB | 1QKI, 2BH9, 2BHL | |
|
GENATLAS • GeneTests • GoPubmed • HCOP • H-InvDB • Treefam • Vega | ||
| Liens accessibles depuis GeneCards et HUGO. | ||
La glucose-6-phosphate déshydrogénase (G6PD) est une oxydoréductase qui catalyse la réaction :
Cette enzyme est la première de la voie des pentoses phosphates, dont elle est l'étape limitante et dont elle contrôle le flux. Elle catalyse l'oxydation du glucose-6-phosphate en 6-phosphoglucono-δ-lactone avec réduction concomitante d'une molécule de NADP+ en NADPH. Elle constitue l'une des sources importantes de ce cofacteur cellulaire.
Un déficit en glucose-6-phosphate déshydrogénase est appelé favisme. Il se caractérise par un déficit en production de NADPH qui est indispensable à la capacité de régénération du glutathion et à la capacité de résister au stress oxydant.
Glucose-6-phosphate déshydrogénase
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
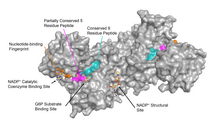
Site de liaison au NAD de la G6PD
Glucose-6-phosphate déshydrogénase de Leuconostoc mesenteroides
| Pfam | PF00479 |
|---|---|
| Clan Pfam | CL0063 |
| InterPro | IPR022674 |
| PROSITE | PDOC00067 |
| SCOP | 1dpg |
| SUPERFAMILY | 1dpg |
Notes et références
[modifier | modifier le code]- (en) Shannon W. N. Au, Sheila Gover, Veronica M. S. Lam, Margaret J. Adams, « Human glucose-6-phosphate dehydrogenase: the crystal structure reveals a structural NADP+ molecule and provides insights into enzyme deficiency », Structure, vol. 8, no 3, , p. 293-303 (PMID 10745013, DOI 10.1016/S0969-2126(00)00104-0, lire en ligne)
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.